7.4 Junção de tabelas utilizando funções4
O pacote base do R fornece uma função que executa essa ação, chamada merge().
Porém, há alguns tipos de junções não podem ser executados com esta função, o que nos levará ao uso de vetores lógicos em conjunto com a função interaction().
Daremos exemplos com essas duas novas maneiras.
7.4.1 Dados para nossa prática
Utilizaremos três tabelas para esta prática:
- O
data.frametab1possui nomes de famílias, gêneros e epítetos específicos de algumas angiospermas:
familia <- c("Burseraceae", "Solanaceae", "Sapindaceae", "Rubiaceae", "Lauraceae")
generos <- c("Protium", "Trattinnickia", "Dacryodes", "Duckeodendron", "Markea", "Solanum", "Allophylastrum", "Cupania", "Thinouia", "Psychotria", "Duroia", "Cinchona", "Ocotea", "Licaria", "Rhodostemonodaphne", "Anisophyllea", "Freziera")
epitetos <- c("aracouchini", "burserifolia", "edilsonii", "cestroides", "ulei", "cyathophorum", "frutescens", "rubiginosa", "myriantha", "viridis", "eriopila", "amazonica", "delicata", "aureosericea", "recurva", "manausensis", "carinata")
tab1 <- data.frame(familia = c(rep(familia, each = 3), "Anisophylleaceae", "Pentaphylacaceae"), genero = generos, epiteto = epitetos, stringsAsFactors = FALSE)| familia | genero | epiteto |
|---|---|---|
| Burseraceae | Protium | aracouchini |
| Burseraceae | Trattinnickia | burserifolia |
| Burseraceae | Dacryodes | edilsonii |
| Solanaceae | Duckeodendron | cestroides |
| Solanaceae | Markea | ulei |
| Solanaceae | Solanum | cyathophorum |
| Sapindaceae | Allophylastrum | frutescens |
| Sapindaceae | Cupania | rubiginosa |
| Sapindaceae | Thinouia | myriantha |
| Rubiaceae | Psychotria | viridis |
| Rubiaceae | Duroia | eriopila |
| Rubiaceae | Cinchona | amazonica |
| Lauraceae | Ocotea | delicata |
| Lauraceae | Licaria | aureosericea |
| Lauraceae | Rhodostemonodaphne | recurva |
| Anisophylleaceae | Anisophyllea | manausensis |
| Pentaphylacaceae | Freziera | carinata |
- O
data.frametab2contem um conjunto pequeno com alguns nomes de famílias, gêneros, e o nome de seus respectivos clados acima dos nomes de ordens segundo o APG (2016):
familia2 <- c("Burseraceae", "Solanaceae", "Sapindaceae", "Rubiaceae", "Annonaceae")
generos2 <- c("Protium", "Duckeodendron", "Thinouia", "Psychotria", "Guatteria")
clado <- c("Malvids", "Lamiids", "Malvids", "Lamiids", "Magnoliids")
tab2 <- data.frame(familia = familia2, genero = generos2, clado = clado, stringsAsFactors = FALSE)| familia | genero | clado |
|---|---|---|
| Burseraceae | Protium | Malvids |
| Solanaceae | Duckeodendron | Lamiids |
| Sapindaceae | Thinouia | Malvids |
| Rubiaceae | Psychotria | Lamiids |
| Annonaceae | Guatteria | Magnoliids |
- O
data.frametab3corresponde à tabela 2,tab2, sem as famílias Solanaceae e Rubiaceae:
| familia | genero | epiteto | |
|---|---|---|---|
| 1 | Burseraceae | Protium | aracouchini |
| 2 | Burseraceae | Trattinnickia | burserifolia |
| 3 | Burseraceae | Dacryodes | edilsonii |
| 7 | Sapindaceae | Allophylastrum | frutescens |
| 8 | Sapindaceae | Cupania | rubiginosa |
| 9 | Sapindaceae | Thinouia | myriantha |
7.4.2 Maneira 3 - função merge()
O básico para entender a função merge() é saber que existem dois argumentos, x e y, que correspondem aos data.frames de entrada.
Quando unimos tabelas, existem junções que adicionam variáveis, e junções que filtram variáveis.
Vamos ver abaixo 4 tipos da primeira ( junção interna, junção à esquerda, junção à direita, junção total), e dois tipos desta última ( semijunção e antijunção).
7.4.2.1 Junção interna
Ao juntarmos tabelas x e y, temos todas as
linhas de x em que há valores em comum com y,
e todas as colunas de x e y. Se houver
múltiplas correspondências entre x e
y, todas as combinações retornam.
Em nosso exemplo, vamos unir as tabelas 1 e 2.
Ambas possuem em comum os identificadores familia e genero.
Para facilitar o entendimento, vamos verificar primeiro cada tabela com cores para checar as correspondências entre x e y nas variáveis em comum:
| familia | genero | epiteto |
|---|---|---|
| Burseraceae | Protium | aracouchini |
| Burseraceae | Trattinnickia | burserifolia |
| Burseraceae | Dacryodes | edilsonii |
| Solanaceae | Duckeodendron | cestroides |
| Solanaceae | Markea | ulei |
| Solanaceae | Solanum | cyathophorum |
| Sapindaceae | Allophylastrum | frutescens |
| Sapindaceae | Cupania | rubiginosa |
| Sapindaceae | Thinouia | myriantha |
| Rubiaceae | Psychotria | viridis |
| Rubiaceae | Duroia | eriopila |
| Rubiaceae | Cinchona | amazonica |
| Lauraceae | Ocotea | delicata |
| Lauraceae | Licaria | aureosericea |
| Lauraceae | Rhodostemonodaphne | recurva |
| Anisophylleaceae | Anisophyllea | manausensis |
| Pentaphylacaceae | Freziera | carinata |
| familia | genero | clado |
|---|---|---|
| Burseraceae | Protium | Malvids |
| Solanaceae | Duckeodendron | Lamiids |
| Sapindaceae | Thinouia | Malvids |
| Rubiaceae | Psychotria | Lamiids |
| Annonaceae | Guatteria | Magnoliids |
Reparem que os valores em que há correspondência entre x e y estão coloridos de amarelo; para os em que não há correspondência, estão coloridos de vermelho.
Agora, executemos a junção das duas tabelas:
| familia | genero | epiteto | clado |
|---|---|---|---|
| Burseraceae | Protium | aracouchini | Malvids |
| Rubiaceae | Psychotria | viridis | Lamiids |
| Sapindaceae | Thinouia | myriantha | Malvids |
| Solanaceae | Duckeodendron | cestroides | Lamiids |
Vejam que houve a incorporação dos valores da coluna epiteto, presente apenas na tabela 2, em que há correspondência entre as tabelas 1 e 2.
É importante notar que as famílias Lauraceae, Anisophylleaceae, e Pentaphylacaceae ficaram de fora, pois não são encontradas na tabela y, isto é, a tabela 2, assim como seus respectivos gêneros e epítetos associados a estes.
Gêneros presentes na tabela 1 de famílias em comum entre ambas as tabelas também não foram incorporados nessa junção, pois nãp encontram correspondência na tabela 2: Dacryodes, Trattinnickia, Markea, Solanum, Allophylastrum, Cupania, Duroia, Cinchona.
Revejam o conceito de junção interna para entender o porquê desse acontecimento.
7.4.2.2 Junção à esquerda
Ao juntarmos tabelas x e y, temos todas as
linhas de x, e todas as colunas de x e
y. Linhas em x sem correspência em
y terão valores NA adicionados nas novas
colunas. Se houver múltiplas correspondências entre
x e y, todas as combinações retornam.
Continuaremos utilizando as tabelas 1 e 2.
Como mostrado anteriormente, ambas possuem em comum os identificadores familia e genero.
Chequemos novamente as cores das correspondências dentro de cada identificador, coloridas em amarelo:
| familia | genero | epiteto |
|---|---|---|
| Burseraceae | Protium | aracouchini |
| Burseraceae | Trattinnickia | burserifolia |
| Burseraceae | Dacryodes | edilsonii |
| Solanaceae | Duckeodendron | cestroides |
| Solanaceae | Markea | ulei |
| Solanaceae | Solanum | cyathophorum |
| Sapindaceae | Allophylastrum | frutescens |
| Sapindaceae | Cupania | rubiginosa |
| Sapindaceae | Thinouia | myriantha |
| Rubiaceae | Psychotria | viridis |
| Rubiaceae | Duroia | eriopila |
| Rubiaceae | Cinchona | amazonica |
| Lauraceae | Ocotea | delicata |
| Lauraceae | Licaria | aureosericea |
| Lauraceae | Rhodostemonodaphne | recurva |
| Anisophylleaceae | Anisophyllea | manausensis |
| Pentaphylacaceae | Freziera | carinata |
| familia | genero | clado |
|---|---|---|
| Burseraceae | Protium | Malvids |
| Solanaceae | Duckeodendron | Lamiids |
| Sapindaceae | Thinouia | Malvids |
| Rubiaceae | Psychotria | Lamiids |
| Annonaceae | Guatteria | Magnoliids |
Em uma junção à esquerda, todas as linhas de x retornam após a junção.
Para executar este tipo de junção, acrescentaremos um novo argumento, all.x = TRUE, indicando que manteremos todas as linhas de x, isto é, o data.frame à esquerda, que é a tabela 1.
| familia | genero | epiteto | clado |
|---|---|---|---|
| Anisophylleaceae | Anisophyllea | manausensis | NA |
| Burseraceae | Dacryodes | edilsonii | NA |
| Burseraceae | Protium | aracouchini | Malvids |
| Burseraceae | Trattinnickia | burserifolia | NA |
| Lauraceae | Licaria | aureosericea | NA |
| Lauraceae | Ocotea | delicata | NA |
| Lauraceae | Rhodostemonodaphne | recurva | NA |
| Pentaphylacaceae | Freziera | carinata | NA |
| Rubiaceae | Cinchona | amazonica | NA |
| Rubiaceae | Duroia | eriopila | NA |
| Rubiaceae | Psychotria | viridis | Lamiids |
| Sapindaceae | Allophylastrum | frutescens | NA |
| Sapindaceae | Cupania | rubiginosa | NA |
| Sapindaceae | Thinouia | myriantha | Malvids |
| Solanaceae | Duckeodendron | cestroides | Lamiids |
| Solanaceae | Markea | ulei | NA |
| Solanaceae | Solanum | cyathophorum | NA |
Agora, temos uma nova situação. Para os valores de x sem correspondência em y, valores NA são acrescentados.
Reparem na coluna clado e vejam que isso ocorreu apenas nesta variável.
Por exemplo, vejam a família Anisophylleaceae. Ela ocorre apenas na tabela 1 e, portanto, não possui nenhum valor de cladoa ssociado a ela, pois esta variável ocorre apenas na tabela 2. Com a junção das tabelas, essa variável é retida, porém sem a existência de um valor para a família, é inserido então o valor NA.
Temos também o caso de Annonaceae, presente na tabela 2. A família não é recuperada na junção interna, pois ela não existe na tabela 1 dentro da variável familia e, portanto, não apresenta correspondência com nenhum dado da tabela 1.
Revejam o conceito de junção à esquerda para entender o porquê desse acontecimento.
7.4.2.3 Junção à direita
Ao juntarmos tabelas x e y, temos todas as
linhas de y, e todas as colunas de x e
y.Linhas em y sem correspência em
x terão valores NA adicionados nas novas colunas. Se houver
múltiplas correspondências entre x e
y, todas as combinações retornam.
De maneira oposta à junção à esquerda, na junção à direita são mantidas todas as linhas de y.
Desta vez, o argumento a ser utilizado é all.y = TRUE.
Antes de executar a junção, vamos checar novamente as variáveis em comum e correspondências entre as tabelas x e y:
| familia | genero | epiteto |
|---|---|---|
| Burseraceae | Protium | aracouchini |
| Burseraceae | Trattinnickia | burserifolia |
| Burseraceae | Dacryodes | edilsonii |
| Solanaceae | Duckeodendron | cestroides |
| Solanaceae | Markea | ulei |
| Solanaceae | Solanum | cyathophorum |
| Sapindaceae | Allophylastrum | frutescens |
| Sapindaceae | Cupania | rubiginosa |
| Sapindaceae | Thinouia | myriantha |
| Rubiaceae | Psychotria | viridis |
| Rubiaceae | Duroia | eriopila |
| Rubiaceae | Cinchona | amazonica |
| Lauraceae | Ocotea | delicata |
| Lauraceae | Licaria | aureosericea |
| Lauraceae | Rhodostemonodaphne | recurva |
| Anisophylleaceae | Anisophyllea | manausensis |
| Pentaphylacaceae | Freziera | carinata |
| familia | genero | clado |
|---|---|---|
| Burseraceae | Protium | Malvids |
| Solanaceae | Duckeodendron | Lamiids |
| Sapindaceae | Thinouia | Malvids |
| Rubiaceae | Psychotria | Lamiids |
| Annonaceae | Guatteria | Magnoliids |
Agora executaremos a junção com o comando abaixo.
Não deixem de reparar no uso do argumento all.y = TRUE, pois ele é o responsável por agora manter todas as linhas da tabela 2 (== y):
| familia | genero | epiteto | clado |
|---|---|---|---|
| Annonaceae | Guatteria | NA | Magnoliids |
| Burseraceae | Protium | aracouchini | Malvids |
| Rubiaceae | Psychotria | viridis | Lamiids |
| Sapindaceae | Thinouia | myriantha | Malvids |
| Solanaceae | Duckeodendron | cestroides | Lamiids |
Notem que agora todos os dados da tabela 2 foram mantidos.
Houve a inserção de um valor NA para a família Annonaceae na variável epiteto, pois esta variável não está presente na tabela 2.
Revejam o conceito de junção à direita para entender o porquê desse acontecimento.
7.4.2.4 Junção total
Ao juntarmos tabelas x e y, temos todas as
linhas e colunas de x e y. Onde não houver
valores correspondentes, valores NA serão colocados nesses
lugares.
Em uma junção total, uniremos todas as linha de x e y utilizando o argumento all = TRUE.
| familia | genero | epiteto | clado |
|---|---|---|---|
| Anisophylleaceae | Anisophyllea | manausensis | NA |
| Annonaceae | Guatteria | NA | Magnoliids |
| Burseraceae | Dacryodes | edilsonii | NA |
| Burseraceae | Protium | aracouchini | Malvids |
| Burseraceae | Trattinnickia | burserifolia | NA |
| Lauraceae | Licaria | aureosericea | NA |
| Lauraceae | Ocotea | delicata | NA |
| Lauraceae | Rhodostemonodaphne | recurva | NA |
| Pentaphylacaceae | Freziera | carinata | NA |
| Rubiaceae | Cinchona | amazonica | NA |
| Rubiaceae | Duroia | eriopila | NA |
| Rubiaceae | Psychotria | viridis | Lamiids |
| Sapindaceae | Allophylastrum | frutescens | NA |
| Sapindaceae | Cupania | rubiginosa | NA |
| Sapindaceae | Thinouia | myriantha | Malvids |
| Solanaceae | Duckeodendron | cestroides | Lamiids |
| Solanaceae | Markea | ulei | NA |
| Solanaceae | Solanum | cyathophorum | NA |
Reparem que valores NA são colocados nos valores da tabela 2 referentes à coluna epiteto, ausente na tabela 1.
O mesmo se passa com valores da coluna clado, presente na tabela 2 e ausente na tabela 1.
Revejam o conceito de junção total para entender o porquê desse acontecimento.
7.4.2.5 Semijunção
Ao juntarmos tabelas x e y, temos todas as
linhas de x onde houver valores correspondentes em
y, mantendo apenas colunas de x. É parecida
com a junção interna, porém difere desta por nunca duplicar valores de
x, retornando sempre apenas valores de x que
houver uma correspondência em y.
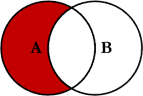
A semijunção é muito similar à junção interna, diferindo desta por não incorporar as colunas de y, pois apenas utiliza esta tabela para filtrar os dados de x, constituindo-se então em um tipo de junção que filtra variáveis.
Neste exemplo, utilizaremos as tabelas 1 e 3.
Ambas compartilham as colunas familia e genero.
Vamos checar primeiramente cada tabela e ver o que é compartilhado entre cada uma:
| familia | genero | epiteto |
|---|---|---|
| Burseraceae | Protium | aracouchini |
| Burseraceae | Trattinnickia | burserifolia |
| Burseraceae | Dacryodes | edilsonii |
| Solanaceae | Duckeodendron | cestroides |
| Solanaceae | Markea | ulei |
| Solanaceae | Solanum | cyathophorum |
| Sapindaceae | Allophylastrum | frutescens |
| Sapindaceae | Cupania | rubiginosa |
| Sapindaceae | Thinouia | myriantha |
| Rubiaceae | Psychotria | viridis |
| Rubiaceae | Duroia | eriopila |
| Rubiaceae | Cinchona | amazonica |
| Lauraceae | Ocotea | delicata |
| Lauraceae | Licaria | aureosericea |
| Lauraceae | Rhodostemonodaphne | recurva |
| Anisophylleaceae | Anisophyllea | manausensis |
| Pentaphylacaceae | Freziera | carinata |
| familia | genero | epiteto | |
|---|---|---|---|
| 1 | Burseraceae | Protium | aracouchini |
| 2 | Burseraceae | Trattinnickia | burserifolia |
| 3 | Burseraceae | Dacryodes | edilsonii |
| 7 | Sapindaceae | Allophylastrum | frutescens |
| 8 | Sapindaceae | Cupania | rubiginosa |
| 9 | Sapindaceae | Thinouia | myriantha |
7.4.2.5.1 Maneira 4 - vetores lógicos e a função interaction()
Para executar uma semijunção com o pacote base do R, devemos fazer uso de vetores lógicos e da função interaction(), pois a função merge() não fornece uma maneira de se obter o que desejamos.
Vamos então à prática5.
As colunas compartilhadas por ambas as tabelas serão nossas chaves:
Partimos então para filtrar na tabela 1 a combinação de linhas para esse conjunto de colunas utilizando a função interaction() do pacote base do R:
## [1] Burseraceae.Protium Burseraceae.Trattinnickia
## [3] Burseraceae.Dacryodes Solanaceae.Duckeodendron
## [5] Solanaceae.Markea Solanaceae.Solanum
## [7] Sapindaceae.Allophylastrum Sapindaceae.Cupania
## [9] Sapindaceae.Thinouia Rubiaceae.Psychotria
## [11] Rubiaceae.Duroia Rubiaceae.Cinchona
## [13] Lauraceae.Ocotea Lauraceae.Licaria
## [15] Lauraceae.Rhodostemonodaphne Anisophylleaceae.Anisophyllea
## [17] Pentaphylacaceae.Freziera
## 119 Levels: Anisophylleaceae.Allophylastrum ... Solanaceae.TrattinnickiaEssa função computa um vetor de fatores que representa a interação das colunas fornecidas na tabela 1. Se fizermos isso com a tabela 3, poderemos saber quais combinações ocorrem em ambas as tabelas.
## [1] Burseraceae.Protium Burseraceae.Trattinnickia
## [3] Burseraceae.Dacryodes Sapindaceae.Allophylastrum
## [5] Sapindaceae.Cupania Sapindaceae.Thinouia
## 12 Levels: Burseraceae.Allophylastrum ... Sapindaceae.TrattinnickiaAgora utilizamos a mesma função interaction e o operador %in% para retornar um vetor lógico que utilizaremos para filtrar os valores da tabela 1 com correspondência na tabela 3.
## [1] TRUE TRUE TRUE FALSE FALSE FALSE TRUE TRUE TRUE FALSE FALSE FALSE
## [13] FALSE FALSE FALSE FALSE FALSE| familia | genero | epiteto | |
|---|---|---|---|
| 1 | Burseraceae | Protium | aracouchini |
| 2 | Burseraceae | Trattinnickia | burserifolia |
| 3 | Burseraceae | Dacryodes | edilsonii |
| 7 | Sapindaceae | Allophylastrum | frutescens |
| 8 | Sapindaceae | Cupania | rubiginosa |
| 9 | Sapindaceae | Thinouia | myriantha |
7.4.2.6 Antijunção
Retorna todas as linhas de x em que não há
correspondência em y, mantendo apenas colunas de
x.
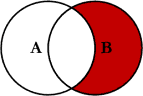
Uma antijunção é ligeiramente diferente de uma semijunção pois ela retorna todas as linhas de x que não aparecem em y.
Portanto, podemos utilizar o inverso de nosso vetor lógico linhas e utilizar este inverso para filtrar as linhas da tabela 1 e ter nossa tabela antijunção entre x e y:
| familia | genero | epiteto | |
|---|---|---|---|
| 4 | Solanaceae | Duckeodendron | cestroides |
| 5 | Solanaceae | Markea | ulei |
| 6 | Solanaceae | Solanum | cyathophorum |
| 10 | Rubiaceae | Psychotria | viridis |
| 11 | Rubiaceae | Duroia | eriopila |
| 12 | Rubiaceae | Cinchona | amazonica |
| 13 | Lauraceae | Ocotea | delicata |
| 14 | Lauraceae | Licaria | aureosericea |
| 15 | Lauraceae | Rhodostemonodaphne | recurva |
| 16 | Anisophylleaceae | Anisophyllea | manausensis |
| 17 | Pentaphylacaceae | Freziera | carinata |
Referências
Texto publicado originalmente no blog de R.O.Perdiz (https://www.ricardoperdiz.com/blog/2020-04-juncao-tbl/)↩︎
Esta solução de semijunção é baseada no tutorial do pacote poorman, recém-criado para emular as funções do pacote dplyr.↩︎